不同的个体对饮食、益生元和益生菌干预往往会表现出不同的反应。越来越多的证据表明,肠道微生物是这种个体异质性的一个关键决定因素。正如每个人都有一个独特的基因型一样,每个人都有一套独特的共生肠道微生物,具有个人特定的功能能力。

这里介绍近期发表在《高级营养学》的观点,这篇文章概述了一些主要的计算和实验工具,这些工具被应用于肠道微生物介导的个性化营养和健康的关键问题。包含四部分主要内容:
-
讨论菌群-营养-健康轴的计算建模的最新进展,包括统计学和人工智能模型的应用。
-
讨论评估个体间异质性的高通量体外技术,从粪便的体外批量培养和厌氧生物反应器中的连续培养,到整合宿主和微生物区间的更复杂的器官芯片模型。
-
探索体内方法,以更好地了解个性化的、由菌群介导的对饮食、益生菌和益生元的反应,从非人类动物模型和人类观察研究,到人类喂养试验和交叉干预。
-
提出了精准营养和医疗保健的未来愿景,即利用肠道菌群来设计有效的、针对个人的干预措施。
下表总结了上述内容中方法的优势和挑战,以及所涉及到的参考文献。
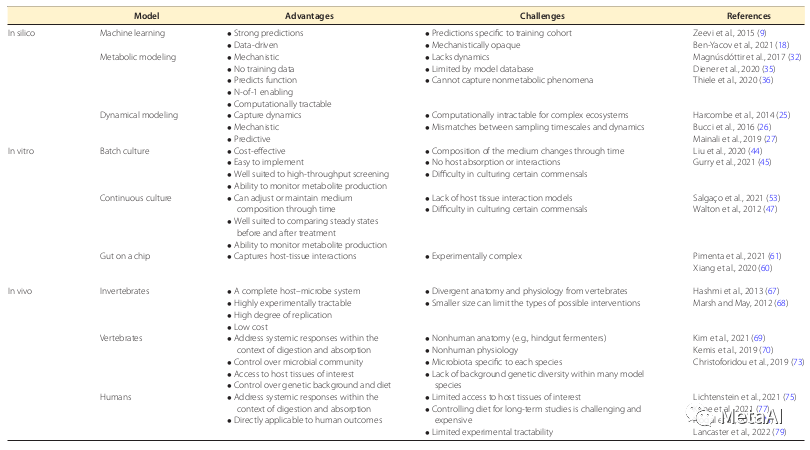
第一部分:计算方法
计算模拟的方法分为2大类,即统计建模和机器学习。
统计建模,比如有多项研究将经典的单变量统计模型应用于接受饮食或生活方式干预的人类队列,并确定了肠道微生物组中具体分类群和功能特征与减肥干预的“反应者”和 “非反应者”之间的关系(见原文中的参考文献10,11,17,后面原文参考直接用数字表示)。此外,基于横断面的相关分析已被用于确定肠道细菌分类群丰度和个性化血脂谱之间的一致关联,以应对饮食的变化(12)。
机器学习(ML)模型对来自个人的多变量表型输入数据(包括肠道微生物组的组成数据)进行训练,已经成功预测了饮食变化的个性化血糖反应,这些个性化的饮食预测最近被证明在管理糖尿病前期的血糖浓度方面优于标准的地中海饮食(9, 18)。
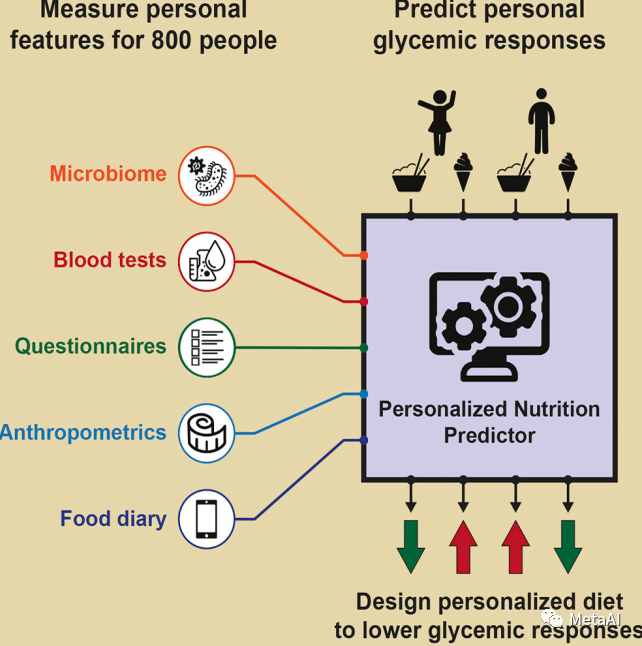
参考文献9中微生物组用于预测血糖
还有其他ML模型,比如根据肠道微生物群的基线组成预测对药物或癌症免疫疗法的个性化反应(13, 19-21)。但这些ML模型的泛化能力依然很差,因此,所得到的模型在训练数据中没有得到很好体现的队列上可能表现不佳。例如,大多数微生物组研究是在美国和欧洲的个体上进行的,这意味着在这些人群中训练的精确干预ML模型在世界其他地区的人群中可能表现不佳(22)。关于ML及其在微生物组介导的精准干预中的应用的更详细回顾,请参见参考文献20、23和24中的评论文章。
还有同时纳入横断面数据和纵向数据的动态模型(dynamic models),用来模拟观察肠道菌群的动态变化。但这类模型参数数量太大,会变得计算困难,需要长时间、高密度的时间序列来进行参数拟合和验证预测(25-27)。跟上述纯横断面的ML模型一样,这类模型的输入数据集往往富含来自发达国家较富裕人群的健康个体,这可能限制了它们对不健康个体、发展中国家的个体或本土社会的应用,因为在这些地方已经观察到了肠道生态学的重大差异(29-31)。
机制模型(mechanistic models )提供了对肠道微生物群和人类健康之间潜在因果关系的详细见解。这类模型不需要训练数据集,因为它们依赖于经过验证的、因果关系的网络。因此,当应用于不同人群时,机制模型可以比统计模型表现得更加稳健。然而,这些模型受到现有知识库中可用信息的限制。例如,我们还没有足够大的关于免疫-共生相互作用的知识库来为这个特定的界面建立一个可靠的机制模型。
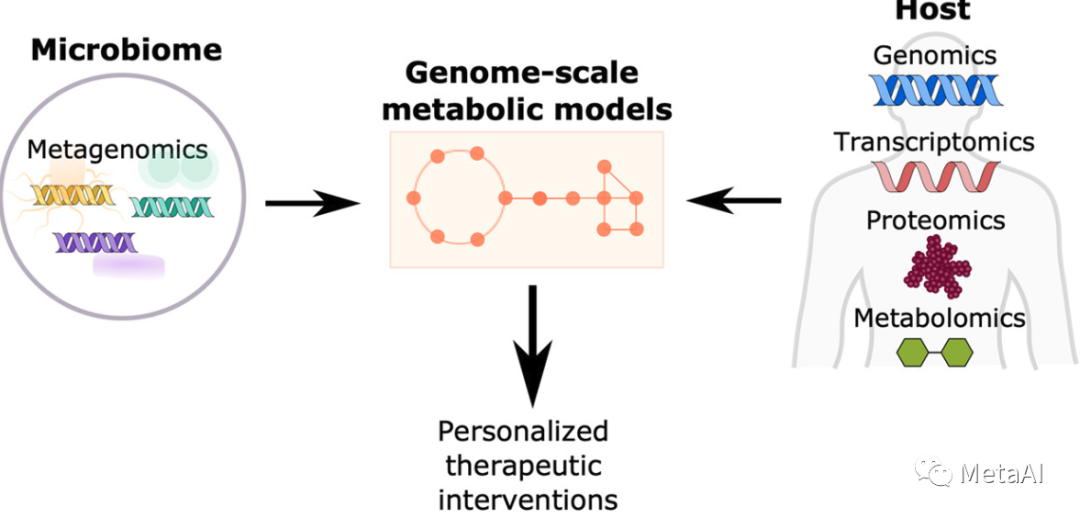
基因组尺度的代谢模型是目前宿主-微生物系统中最有希望的一种机制模型,因为有大量关于人类和肠道细菌代谢的已有知识(32, 33)。先前的工作表明,肠道中的基线微生物代谢基因谱可用于预测益生菌菌株的移植概率,表明当益生菌的代谢位被其他已建立的共生菌占据时,它就无法移植(34)。
最近在将多种人类肠道细菌共生体整合到一个可计算的宏基因组尺度的代谢模型中取得了进展,该模型可以将生态系统结构与代谢产出进行映射,并允许对饮食、益生元和益生菌干预的代谢反应进行个性化模拟(35)。此外,在过去几年中,在将宿主和肠道细菌代谢整合到整个身体代谢模型方面取得了重大进展(36)。这些类型的模型能够在宿主代谢变化的背景下模拟肠道共生菌的代谢,如苯丙酮尿症或成年后乳糖酶表达的丧失(8,36)。事实上,生态系统规模的代谢模型是构建“数字双胞胎(digital twin)”(即个人的计算模型,可用于在计算模拟中评估潜在干预措施的影响)的有力工具,用于设计和测试模拟中的益生元、益生菌和饮食干预措施(8, 37,关于微生物组代谢模型和精准医疗的更全面的回顾,见参考文献8)。然而,新陈代谢只是更广泛的一个部分,仍然需要将这些模型与免疫系统、神经系统、内分泌系统和行为的模型相结合(37)。积累高质量的纵向数据,使用标准化的方法从人类群体中收集,对于这些机制化的宿主-微生物群模型的发展和验证至关重要。
总的来说,当涉及到肠道微生物群的精确工程以优化健康时,需要统计学和机制学方法来取得进展。数据密集型统计方法是对宿主-微生物群界面进行预测的最佳方法,但这些模型由于其性质,往往在解释上有些不透明。另一方面,机制模型利用现有的和新出现的知识库以及对微生物群组成和饮食的个性化约束来进行个性化的N-of-1预测,不需要训练数据,并且有更透明的、可解释的生物学输出。这类N-of-1,或 “数字双胞胎”的个性化模型,能够进行计算精确干预反应预测,在其他领域(如癌症或传染病的治疗)越来越受欢迎(38,39)。
(未完待续)
参考文献:
Gibbons SM, Gurry T, Lampe JW, Chakrabarti A, Dam V, Everard A, Goas A, Gross G, Kleerebezem M, Lane J, Maukonen J, Penna ALB, Pot B, Valdes AM, Walton G, Weiss A, Zanzer YC, Venlet NV, Miani M. Perspective: Leveraging the Gut Microbiota to Predict Personalized Responses to Dietary, Prebiotic, and Probiotic Interventions. Adv Nutr. 2022 Oct 2;13(5):1450-1461. doi: 10.1093/advances/nmac075. PMID: 35776947; PMCID: PMC9526856.
|